In Eindhoven is bedacht hoe je synthetische cellen zo kunt bouwen dat ze onderling DNA-strengen kunnen uitwisselen. Zo kun je ze uiteindelijk laten werken als ‛DNA-computer’, suggereren Tom de Greef en collega’s in Nature Nanotechnology.
Het idee is dan dat je elke cel van tevoren laadt met twee DNA-strengen die deels op elkaar passen. De input is een derde streng die veel beter past op een van de twee en dus de tweede streng verdringt. Die verlaat dan de cel op weg naar eentje met een andere set DNA, waarin hij op zijn beurt voor derde streng kan spelen.
Om het effect zichtbaar te maken voorzie je de eerste streng van een fluorescerende groep, en de tweede van een groep die de fluorescentie afknijpt zolang beide strengen op elkaar zitten.
Door het iets ingewikkelder te maken kun je allerlei logische schakelingen opbouwen: denk aan constructies waarbij twee verschillende inputstrengen nodig zijn eer er één outputstreng vrij komt. En je kunt het signaal versterken door een vierde streng (fuel strand) toe te voegen die niet de tweede kan verdringen, maar wel de derde - zo blijft de cel in kwestie geactiveerd terwijl die derde streng aan de slag kan in een volgende cel.
Tot zover niets nieuws maar tot nu toe werkten zulke constructies alleen in vitro. Om het DNA toegankelijk te houden presenteerde men het open en bloot, bijvoorbeeld op het oppervlak van een nano- of microdeeltje. En in een ‛biologisch relevant’ medium, zoals bloedserum, blijft DNA dan niet lang goed.
Wat De Greef en collega’s biomolecular implementation of protocellular communication (BIO-PC) noemen komt er in het kort op neer dat je je synthetische ‛protocellen’ voorziet van een celwand waar korte DNA-strengen (zeg 100 basen of minder) makkelijk doorheen kunnen. Daartoe vervang je de gebruikelijke lipides door een soort blokcopolymeer van runderalbumine en poly-N-isopropylacrylamide. In plaats van liposomen levert dat dus proteïnosomen op, in dit geval met diameters van 10 tot 60 µm.
Je zorgt dat het inwendige een hoeveelheid streptavidine bevat. Aan het vrije uiteinde van je DNA hang je biotinemoleculen, die streptavidine binden. De strengen worden dan vanzelf de proteïnosomen ingetrokken. Hoe snel ze er precies in en uit diffunderen, en dus hoe nerveus de ‛computer’ reageert, kun je tamelijk nauwkeurig instellen door met de chemische samenstelling van de wand te spelen.
Het DNA wordt nu alleen nog afgebroken op momenten dat het buiten die protocellen beweegt. En die tijd is zo kort dat het tamelijk lang goed gaat.
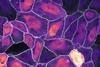
Om er experimenten mee te kunnen doen vervaardig je een soort microrekje waarop je de proteïnosomen op vaste afstanden van elkaar kunt fixeren - zie de lichtgrijze vlekjes op de foto. Die toont een signaalcascade met drie verschillende protocellen die respectievelijk groen, blauw en cyaan oplichten wanneer ze hun tweede streng hebben geloosd, als bewijs dat het principe werkt.
In hoeverre zo’n proteïnosoomconstructie zich daadwerkelijk leent voor synthetische cellen, waarbij de celwand ongetwijfeld veel meer functies heeft dan het doorlaten van DNA, is afwachten. Maar je moet het idee wel degelijk kunnen inzetten voor medische diagnostiek, waarbij je bepaalde stoffen in het bloed de onderlinge communicatie tussen de proteïnosomen laat beïnvloeden. Volgens een persbericht van de TU/e zijn er al contacten met softwarebedrijf Microsoft om dit nader uit te werken.
bron: TU/e, Nature Nanotechnology














Nog geen opmerkingen