Menselijke pluripotente stamcellen kunnen alle soorten cellen worden, maar bezitten epigenetische mechanismen die sommige celpaden tegenhouden, blijkt uit een Vlaams-Nederlandse studie in Nature Cell Biology.
Naïeve menselijke pluripotente stamcellen (hPSC’s) kunnen in theorie net als pre-implantatie morula embryos differentiëren tot vrijwel alle mogelijke soorten celtypes. Maar er zijn mechanismen aanwezig die de hPSC’s begrenzen, ontdekten de groepen van Maarten Dhaenens, Hendrik Marks, Peter Rugg-Gunn en Vincent Pasque.
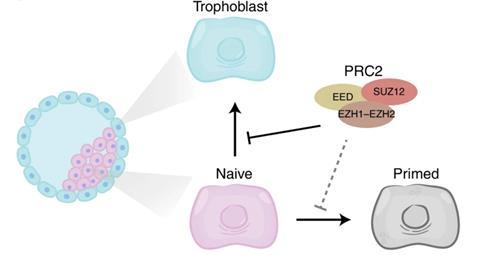
De onderzoekers uit respectievelijk Gent, Nijmegen, Cambridge en Leuven keken specifiek naar de vorming van trofoblast-cellen uit hPSC’s. Trofoblast-cellen ontstaan normaal gesproken vier dagen na de samensmelting van een menselijke zaad- en eicel uit het morula embryo en vormen de basis voor de placenta. Normaliter ontstaan trofoblast-cellen echter niet spontaan uit hPSC’s, wat lijkt te suggereren dat de vorming actief onderdrukt wordt. Met een multi-omics-aanpak onderzochten ze wat die barrière zou kunnen zijn.
‘Omics-technieken zijn studies die een heel panel aan biomoleculen bestudeert’, legt Maarten Dhaenens (UGent) uit. ‘We gebruikten verschillende omics-technologieën (samen ‘multi-omics’, red.) om de eiwitten, het DNA en het RNA te bestuderen in een zo vroeg mogelijk regulatiestadium van de hPSC’s. Daarbij ging onze aandacht vooral naar de epigenetische mechanismen die beslissen welke genen geactiveerd worden om uiteindelijk in eiwitten te worden vertaald. Hierin spelen histonmodificaties en methylaties van DNA een heel belangrijke rol.’
Ons genetisch materiaal is namelijk complexer dan alleen de dubbele DNA-helix. Hendrik Marks (Radboud): ‘DNA zit verpakt rond histonen, en de expressie tot RNA en andere processen wordt gereguleerd door transcriptiefactoren en enzymatische processen. Die laatsten zetten het DNA open voor transcriptie, afhankelijk van of een transcriptiefactor bindt aan het gen en de andere enzymen kan recruteren.’
Rem
Multi-omics kun je ruwweg verdelen tussen twee velden: massaspectrometrie (MS) voor eiwitten enerzijds, en sequencing of aflezen van DNA en RNA anderzijds. ‘Bij de epigenetica komen die twee samen’, vertelt Dhaenens. ‘Met MS zagen we dat een bepaalde histonmodificatie veel meer voorkomt bij stamcellen die een richting hebben gekozen dan bij naïeve stamcellen. Met sequencing hebben we vervolgens gekeken waar op het DNA de bobijntjes (spoelachtige structuren van histonen om het DNA, red.) met die modificatie zich bevonden.’
De histonmodificatie die de onderzoekers heel specifiek in de hPSCs vonden wordt aangebracht door het eiwitcomplex PRC2. Het gaat om een trimethylering op lysine nummer 27 van histon 3, afgekort H3K27me3. ‘Interessant genoeg vonden we dat H3K27me3 op genen zat die belangrijk zijn voor placentaontwikkeling’, vult Marks aan. Die genen bleken uit te staan, wat de placentaontwikkeling dus tegenhoudt. ‘Na die stappen probeerden we PRC2 en dus H3K27me3 te remmen met kleine moleculen. Uit daaropvolgende RNA-sequencing bleek dat de cellen zonder de H3K27me3-rem inderdaad veel efficiënter richting placentacellen gingen.’ Het PRC2-eiwitcomplex fungeert dus als een soort poortwachter.
Tegelijk
Volgens de onderzoekers sluit dit goed aan bij eerdere observaties. ‘We zagen ook hoge H3K27me3 in pluripotente stamcellen van muizen’, zegt Dhaenens. Tegelijk met de huidige studie publiceerde een andere groep over precies hetzelfde onderwerp een paper in hetzelfde tijdschrift. ‘Voor ons is dat heel belangrijk, een teken dat onze bevindingen heel herhaalbaar en robuust zijn. Het geeft ons het gevoel dat we inderdaad de juiste richting in zijn geslagen, het maakt ons zekerder van ons werk.’
‘We hebben mooie data verzameld’, vertelt Marks. ‘Daarbij hebben we veel profijt gehad van de recente ontwikkelingen in embryonale systemen.’ Vorig jaar publiceerde Nicolas Rivron, een van de co-auteurs werkzaam bij een onderzoeksinstituut in Wenen, een nieuwe methode om embryo-achtige structuren te laten groeien. ‘We konden die modellen krachtig gebruiken om onze bevindingen te testen, wat mooie microscopische afbeeldingen opleverde (zie hieronder, red.) die goed de rol van H3K27me3 laat zien.’
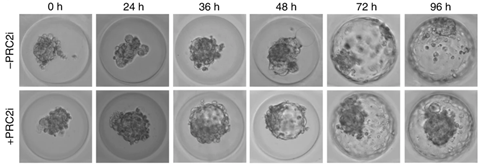
Zijlmans, D.W. et al. (2022) Nat Cell Biol 24, doi.org/10.1038/s41556-022-00932-w














Nog geen opmerkingen