In Groningen heeft een bacterie enzymen aangemaakt met een 21ste aminozuur dat koperionen bindt. In vitro kun je daar Friedel-Crafts-alkyleringen enantioselectief mee katalyseren, schrijven Ivana Drienovská en Gerard Roelfes in het tijdschrift Chemical Science.
Voor zover bekend hebben ze daarmee een wereldprimeur te pakken. In vitro zijn al vaker enzymen gemaakt met synthetische metaalbindende aminozuren. Het inbouwen van onnatuurlijke aminozuren door bijvoorbeeld E.coli is ook vaker gedemonstreerd. Maar een metaalbindend aminozuur in een coli is nieuw.
Die metaalbinding werkt heel anders dan in de natuur. Daar wordt het ion nooit door één enkel aminozuur gebonden, maar klemgezet tussen functionele groepen van een aantal aminozuren tegelijk, eventueel in combinatie met een klein organisch molecuul als cofactor. Dat zo’n ion precies past in de 3D-vouwstructuur van de functionele plek van het eiwit is een kwestie van miljoenen jaren evolutie. Als je niet zo lang de tijd hebt, is een synthetisch metaalbindend aminozuur wel zo handig.
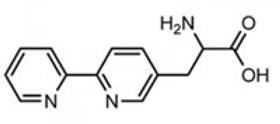
Dat aminozuur in kwestie is (2,2’-bipyridin-5-yl)alanine, afgekort BpyAla. Dit kan Cu2+ binden via de twee stikstofkernen in de pyridineringen. De Groningers bouwden het in via de inmiddels al haast klassieke ‘amber stop codon suppression methodology’, waarbij je één van de drie natuurlijke ‘stopcodons’ uit de genetische code een nieuwe betekenis geeft. Het uitgangsenzym was een transcriptiefactor genaamd LmrR, waar ze BpyAla in monteerden op één van drie plekken die op grond van eerder onderzoek interessant leken.
Uiteindelijk moeten zulke gemodificeerde enzymen iets gaan doen binnen het metabolisme van de producerende bacterie. Maar zo ver zijn ze nog niet. Voorlopig worden de enzymen nog uit de bacteriën geïsoleerd en chromatografisch opgezuiverd, waarna ze in een reageerbuisje met een koper(II)nitraatoplossing mogen laten zien wat ze kunnen.
In dit geval mochten ze de Friedel-Craftsreactie tussen 5-methoxy-1H-indool en 1-(1-methyl-1H-imidazol-2-yl)but-2-en-1-on katalyseren. Van het product bestaan twee enantiomeren en met het eiwit blijk je ze allebei redelijk selectief te kunnen maken. De plek waar BpyAla en us het koper in het eiwit zit, bepaalt hierbij welk enantiomeer je krijgt.
Volgens Roelfes heeft deze aanpak dus het grote voordeel dat je heel snel die plek kunt wijzigen. De benodigde genetische codes zitten niet in het ‘vaste’ DNA van E.coli maar in een los plasmide. Als je met de plek van het metaalion wilt spelen hoef je alleen dat plasmide een beetje te wijzigen en het in de volgende E.coli te monteren - fermentoren genoeg. Uiteindelijk zou je zo via een vorm van ‘gerichte evolutie’ het enzym kunnen optimaliseren.
bron: Chemical Science, ChemistryWorld















Nog geen opmerkingen