Een goed getraind algoritme heeft aan een massaspectrum genoeg om de molecuulstructuur van een nieuwe designer drug te raden.
Vraag niet hoe het kan maar profiteer ervan, suggereert Princeton-onderzoeker Michael Skinnider die met dit algoritme de NOMIS & Science Young Explorer Award 2023 ter waarde van $15.000 in de wacht sleepte.

Zogeheten designer drugs, ook bekend als novel psychoactive substances (NPS), komen voort uit de manier waarop drugswetgeving traditioneel in elkaar zit. Net als bijvoorbeeld bij PFAS zijn alleen moleculen verboden waarvan de structuur exact zo in de wet staat. Wijzig één zijgroep en je hebt iets dat een vergelijkbaar effect heeft op de menselijke hersenen, maar volkomen legaal is totdat de overheid haar zwarte lijst bijwerkt – waarna je dezelfde truc opnieuw kunt uithalen.
Een logische tegenmaatregel (alweer net als bij PFAS) is dat je complete stoffenfamilies verbiedt aan de hand van hun koolstofskelet, ongeacht de zijgroepen. Sommige landen, waaronder België en Duitsland, hebben die stap jaren geleden al gezet. Maar in Nederland wacht het wetsvoorstel nog altijd op behandeling in de Tweede Kamer.
Meestal wordt pas duidelijk dat de drugsmaffia weer iets nieuws heeft verzonnen wanneer gebruikers in het ziekenhuis belanden met ongebruikelijke symptomen. Skinnider noemt als voorbeeld een gemodificeerd cannabinoïde, dat zombiegedrag veroorzaakte. Om de slachtoffers adequaat te kunnen helpen, is het zaak zo snel mogelijk de molecuulstructuur te achterhalen. Massaspectrometrie ligt voor de hand, maar geeft alleen uitsluitsel als je een referentiespectrum kunt vinden. Zo niet, dan moet je een aanvullende NMR-analyse doen die veel bewerkelijker is.
ChatGPT
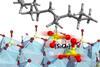
Skinnider vermoedde dat je meer informatie uit een massaspectrum kunt halen met large language models, neurale netwerken die ook de basis vormen van bijvoorbeeld ChatGPT. Met het SMILES-protocol (simplified molecular-input line-entry system) digitaliseerde hij een database van 1753 bekende NPS-structuren. Om zo’n AI te trainen zijn dat er veel te weinig, maar Skinnider maakte handig gebruik van het feit dat je een SMILES-vertaling kunt laten beginnen bij een atoom naar keuze en dan telkens een andere omschrijving van dezelfde structuur krijgt.
Het getrainde model mocht daarna nieuwe designer drugs bedenken. Daar kwamen miljoenen moleculen uit die aardig leken te voldoen aan de functie-eisen, zoals synthetiseerbaarheid en potentieel om door de bloed-hersenbarrière te dringen. Maar verrassend genoeg doken de meeste maar één- of tweemaal op in de output, terwijl andere tienduizenden keren opnieuw werden gesuggereerd.
Hoe dat komt is een raadsel maar Skinniders gok dat de logica van zijn AI overeenkomt met die van de drugsmaffia lijkt er niet ver naast te zitten. Van 194 NPS’en, die nadien zijn opgedoken, bleken er 176 bij de favoriete suggesties te staan. Sinds Skinnider dit twee jaar geleden publiceerde in Nature Machine Intelligence, heeft zijn AI al minstens één onbekende drug correct geïdentificeerd op basis van alleen de totale molecuulmassa. Neem je ook de overige details van de MS-spectra mee, dan werkt het nog veel beter: zeker veertig structuren van pas ontdekte NPS’en zijn zo al opgehelderd.
Het prijswinnende essay: Michael Skinnider, Hallucinating hallucinogens, Science (2023)
De originele paper: Michael Skinnider, et al., A deep generative model enables automated structure elucidation of novel psychoactive substances, Nature Machine Intelligence (2021)
Meer over de NOMIS & Science Young Explorer Award


















Nog geen opmerkingen