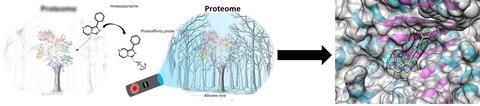
Onderzoekers uit Leuven laten met photoaffinity probes zien dat je de off-targets van een bekend antikankergeneesmiddel dat is gericht op kinases niet alleen terugvindt in kinasefamilies, maar ook in andere eiwitten. Ze publiceerden hun bevindingen in Communications Chemistry.
Een veelgebruikte manier om enzymen te bestuderen is het gebruik van activity-based probes. Deze kleine moleculen bevatten een reactief gedeelte dat het substraat van een enzym nabootst. Afhankelijk van de probe kun je zo een covalente binding vormen tussen de probe en het enzym. Als je een moleculair label aan de probe bevestigt, kun je het enzym er vervolgens uit filteren voor analyse.
‘Maar soms zijn er enzymatische mechanismes die je niet op die manier kunt remmen’, zegt Steven Verhelst, hoogleraar chemische biologie aan de KU Leuven. In dat geval kun je photoaffinity based probes gebruiken. ‘Deze omzeilen het probleem door zich niet-covalent aan het enzym te binden. Een paar jaar geleden begon onze groep naar verschillende doelwitten te kijken, waaronder kinases, om deze methode op toe te passen.’
Rond die tijd trok Verhelst ook Dimitris Korovesis aan als postdoc, die al ervaring had met de synthese en toepassing van kinaseremmers en imaging probes. Wanneer een kinase niet goed functioneert kan dat leiden tot verschillende soorten kanker, waarvoor kinaseremmers als medicijn kunnen dienen. ‘Maar zoals met alle medicijnen wil je weten hoe specifiek ze zijn en of er andere eiwitten zijn waar ze zich op richten’, zegt Korovesis, die nu postdoctoraal onderzoeker is aan het Instituut voor Moleculaire Biologie en Biotechnologie op Kreta.
Triviaal
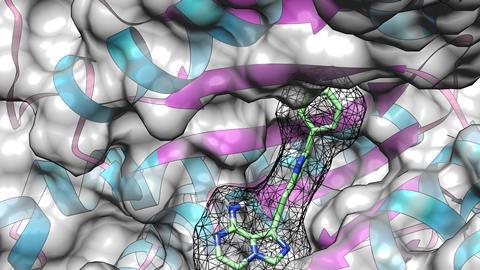
Korovesis begon met een experimenteel molecuul – KIRA6 – dat was ontworpen om een specifiek eiwit, IRE1α, aan te pakken. Een andere groep wist dat het molecuul een off-target had, maar ze wisten niet welke. ‘We vonden wat ze zochten, maar wat ons daarbij opviel was dat KIRA6 een structurele kern heeft die overeenkomt met verschillende andere kinaseremmers.’
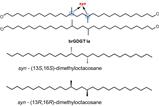
Toen vroegen Korovesis en Verhelst zich af: welke kleine verschillen maken deze zeer vergelijkbare moleculen selectiever? Met deze vraag in gedachten selecteerden ze een paar bekende kinaseremmers en synthetiseerden ze er zelf ook een aantal, allemaal met dezelfde imidazopyrazine-kernstructuur, om ze vervolgens allemaal te voorzien van photoaffinity-labels om hun selectiviteit te bekijken.
Tijdens de synthese, die deels werd uitgevoerd door masterstudent Christel Mérillat, kwam het team een aantal hordes en tegenslagen tegen. ‘We moesten meermaals wisselen van beschermgroepen, bijvoorbeeld omdat we zagen dat degene die we hadden gekozen eraf viel tijdens de zuivering’, zegt Korovesis. Een ander voorbeeld is de bescherming van een aromatisch amine in de pyrazinering. ‘Volgens de literatuur zou je die niet hoeven te beschermen om een koppelingsreactie te doen, maar in onze handen wilde het niet werken en moesten we hem dus wel beschermen om de diazirine te maken.’ Verhelst voegt toe: ‘Het kiezen van je beschermgroepen kan soms triviaal lijken, maar deze situatie laat zien dat dat niet altijd het geval is.’
Onvolledig
Het is belangrijk om off-target profielen breder te bekijken bij het ontdekken en ontwikkelen van geneesmiddelen, zegt Verhelst. ‘Je moet niet alleen kijken naar de selectiviteit van kinaseremmers binnen het kinoom [de familie van kinase-enzymen, red.], maar binnen het bredere proteoom. Je kunt de selectiviteit van kinases op verschillende manieren beoordelen, maar sommige daarvan kijken alleen naar het kinoom zelf, waardoor de bepaling van selectiviteit onvolledig is als je niet ook kijkt naar de andere ATP- en nucleotidebindende eiwitten die we als off-targets hebben gevonden.’
Als je deze off-targets in een vroeg stadium vindt, kom je later in het medicijnontwikkelproces niet voor onverwachte verrassingen te staan. ‘En ik wil daarmee niet suggereren dat je per se photoaffinity-labeling moet gebruiken, zolang je maar naar het hele proteoom kijkt. Ik denk dat dat de belangrijkste boodschap van dit werk is’, besluit Verhelst.
Korovesis, D. et al. (2025) Commun Chem 8(34), DOI: 10.1038/s42004-025-01436-y

















Nog geen opmerkingen