Nijmeegse chemici ontwikkelden een reservoir computing systeem, waarbij ze de formosereactie gebruiken om complexe berekeningen te doen, staat in Nature. ‘Je kunt dezelfde experimentele data meerdere keren gebruiken om honderden computationele taken te verrichten.’
‘Al sinds het begin van de computertijd dachten mensen aan het gebruik van moleculen in plaats van transistors in computers, omdat die toentertijd kleiner waren’, vertelt Wilhelm Huck, hoogleraar fysisch organische chemie aan de Radboud Universiteit (RU). ‘Maar tegenwoordig kleven daar nadelen aan: transistoren zijn nu kleiner dan moleculen en een computer op basis van moleculen werkt veel trager dan met elektronen.’
Daarom is het moleculair computeridee overgestapt op DNA, waarbij je met slimme schakelingen in het basenetwerk berekeningen kunt doen. Een tijd geleden lieten onderzoekers bijvoorbeeld zien dat je de wortel van een bepaald getal uit kunt rekenen met een hele serie DNA-reacties.
‘We simuleerden het E. coli-koolstofmetabolisme met ons formose-reservoir’
‘Maar wat wij doen is een heel ander concept, het gebruikt niet de normale computerarchitectuur’, vervolgt Huck. Hij heeft het over reservoir computing (RC), uitgevonden door de van origine Duitse Herbert Jaeger, die nu hoogleraar aan de Rijksuniversiteit Groningen is. RC kijkt naar waar een computer slecht in is, namelijk niet-lineaire processen. ‘Dat komt omdat je hele kleine stapjes moet nemen over een langere tijd om te zien wat er gebeurt’, zegt Huck. ‘En dat wordt snel ongelooflijk duur. Maar zulke processen zou je eigenlijk juist met computers willen bekijken. Denk aan klimaatmodellen of complexe chemische reacties.’ RC biedt daar dus een oplossing voor.
Automatisch
Maar waarom werkt moleculair reservoir computing zo goed? ‘Omdat je de reactie die we gebruiken, de formose-reactie, niet hoeft te trainen’, legt Huck uit. ‘Elke chemische reactie heeft zijn eigen karakteristieken, zijn eigen reactieconstanten. Daardoor gaat de training automatisch.’
Er zijn vier soorten input voor het reservoir, die je via een flowsysteem kunt beïnvloeden: formaldehyde, dihydroxyaceton, NaOH en CaCl2. De output bestaat dan uit massaspectrum, gemeten met een ion mobility MS. Op het spectrum kun je 106 verschillende ionen zien, ‘hoewel niet elk ion uniek is, sommige traces zijn identiek’, aldus Huck. De output is dus afhankelijk van de input en de niet-lineaire respons (de verzameling ionen) kun je vervolgens uitlezen door verschillende ‘gewichten’ te hangen aan de ionsignalen. Met verschillende gewichtensets kun je dezelfde experimentele data vervolgens meerdere keren gebruiken om honderden computationele taken te verrichten.
‘Na een dag metingen doen rook het hele MS-apparaat naar karamel’
Daar gaven Huck en collega’s meteen een goed voorbeeld van. Huck: ‘Het reservoir kan een hoop wiskundige vergelijkingen tegelijk oplossen. Zo deden we een experiment waarbij we een model van het koolstofmetabolisme van een E. coli-bacterie simuleerden met ons formose-reservoir.’ En dat lukte, ondanks dat het metabolisme bestaat uit 87 substraten en 92 reacties, dus een vrij complex biochemisch systeem.
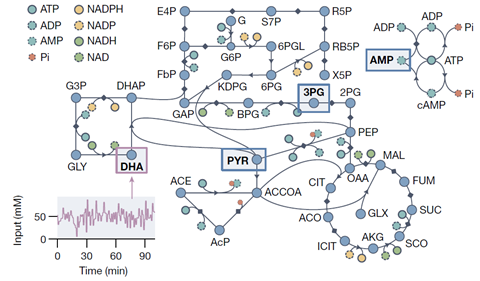
Realisatie
‘Dat dit überhaupt mogelijk is, vind ik heel bijzonder’, zegt Huck. ‘Normaal zie je dit alleen in silico en er zijn enkele voorbeelden in materialen, maar nu blijkt dat dit werkt, opent dit de deur naar een heel grote groep chemische systemen die grote hoeveelheden informatie kunnen verwerken.’
Het zorgde voor een realisatie, vertelt Huck. ‘In een levende cel bevinden zich vele netwerken. Tot nu toe hebben we die netwerken vooral als lineaire paden beschouwd, maar mogelijk laten chemische domeinen als biologische cellen zich veel beter modelleren door zo’n reservoir computing systeem. De informatie die je in het formose reservoir stopt verspreidt zich over alle dimensies van het netwerk, wat betekent dat chemische systemen die een vergelijkbare verknoopte netwerkstructuur hebben ook eigenschappen zullen hebben om informatie te verwerken.’
Bruine zooi
In het begin nam het team samples met een GCMS, waarin je elke paar minuten een spectrum meet. Zo hebben ze duizenden spectra gemeten, maar voor het maken van tijdseries is dit veel te traag. ‘Toen besloten we om onze samples rechtstreeks te injecteren in de massaspectrometer van Jana Roithová [hoogleraar spectroscopie en katalyse, RU, ed.], zonder enige voorbewerking.’
Dat was nogal een risico. ‘De formosereactie gebeurt met suikers en vormt langzaam karamel en uiteindelijk zwarte teer. Na een dag metingen doen rook het hele apparaat dus naar karamel en moeste we die bruine zooi uit het apparaat halen. We dachten dat de MS het niet zou overleven, maar we zijn al een jaar bezig en het blijkt gewoon te kunnen.’
‘Als het kwartje eenmaal gevallen is, zie je overal wel mogelijkheden om reservoir computing toe te passen’
Om dit werk handen en voeten te geven in het veld, werkt de groep van Huck samen met computerbedrijf IBM om zo’n reservoir computing reactor te bouwen in een siliconenchip. ‘Met dezelfde technieken als waarmee je geavanceerde computerchips maakt, zou je ook reactorchips moeten kunnen maken’, legt Huck uit. ‘Wat wij nu doen met pompjes, slangetjes en MS, moet je dan verkleinen naar microliterschaal zodat je tientallen formosereactors op één chip kunt zetten. Op de lange termijn zou je dan serieuze computingproblemen kunnen oplossen.’
Pong
De eigen onderzoeksgroep staat ook niet stil. ‘Intern zijn we bezig met een ander netwerk dat ook werkt, maar met formose gaan we verder naar een meer autonoom systeem, waarbij je data van de MS real time verwerkt en gebruikt om de pompen van je reactor aan te sturen. Een zelf-aandrijvend systeem dus. Promovendus Thijs de Jong werkt daar nu mee en is bijna zo ver dat hij de reservoircomputer Pong tegen zichzelf kan laten spelen, wat bewijst dat de formosereactie echt een krachtige computer is. Als het kwartje eenmaal gevallen is, zie je overal wel mogelijkheden om reservoir computing toe te passen.’
Baltussen, M.G. et al. (2024) Nature, DOI: 10.1038/s41586-024-07567-x




















Nog geen opmerkingen